近日,我校计算机与控制工程学院生物信息学实验室章天骄副教授团队在生物组织空间域识别领域取得重要突破,提出了一种将自编码器与多尺度自适应图卷积网络(MACM)结合,通过动态平衡spot属性特征和空间邻域结构信息进行空间域识别的新方法spaMGCN。相关成果以“spaMGCN: a graph convolutional network with autoencoder for spatial domain identification using multi-scale adaptation” 为题发表于Genome Biology(中科院1区TOP期刊,影响因子9.4)期刊上。
识别空间域是分析空间转录组学数据的关键步骤之一,是理解组织异质性的基础。传统方法大多使用单一组学数据识别空间,然而,单组学数据中包含的信息量是有限的,尤其是在组织结构复杂的情况下,这使得为转录组学数据开发的空间域识别方法难以获得理想的结果。空间多组学测序技术的出现打破了空间转录组学只能捕获单组学数据的传统局限,从而导致空间域鉴定方法从单一转录组学数据应用转向空间多组学数据的集成。在空间多组学数据的分析中,不同组学提供的信息是互补的,允许对组织结构进行多方面的表征。通过整合空间多组学数据,可以克服单组学数据的固有局限性,从而实现更准确的空间域识别。以往的大多数方法在处理连续聚集的组织分布时表现尚可,但对离散分布(如淋巴滤泡)和相邻组织(如脑皮层与头骨)的识别仍存在边界模糊问题,亟需开发新型算法以解决这些问题。
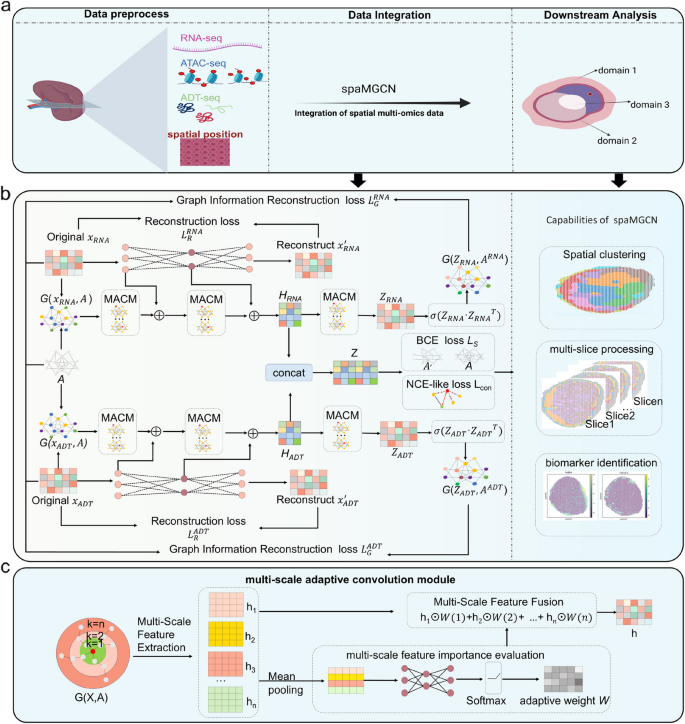
为了突破现有空间组学技术在离散组织识别中的局限性,章天骄副教授团队开发了spaMGCN模型,创新性地将自编码器与多尺度自适应图卷积网络(MACM)结合。该模型通过动态平衡spot属性特征(如转录组/蛋白组)和空间邻域结构信息,在空间域识别领域取得了较大提升。
spaMGCN不仅能精准识别离散分布的同一空间域,还可清晰分离相邻组织结构,同时支持多切片数据并行处理及大规模数据集分析,在单细胞分辨率数据集上也展现出强大适应性。研究团队通过差异分析验证了其识别的空间域与生物功能的强相关性,为发现潜在生物标志物提供新方向。


原文链接:https://doi.org/10.1186/s13059-025-03637-z